3.1 KiB
BZKF Real World Data Platform - Plausibilitätsprüfung
Anwendung zur Durchführung einer Plausibilitätsprüfung anhand der Daten für die BZKF Real World Data Platform.
Aufbau der ETL-Strecke an den Standorten
Die Daten werden aus der Onkostar-Datenbank ausgelesen und in Apache-Kafka eingespeist. Nach Durchlauf der ETL-Strecke wird das Ergebnis in einer CSV-Datei gespeichert. Diese wird dann (aktuell) manuell in OPAL importiert.
flowchart LR
A[Database] --> B[Kafka-Connect]
B --> C[ADT to FHIR]
C --> D[fhir-pseudonymizer]
D --> E[obds-fhir-to-opal]
E -->|CSV-File| F[OPAL]
Ermittelte Kennzahlen
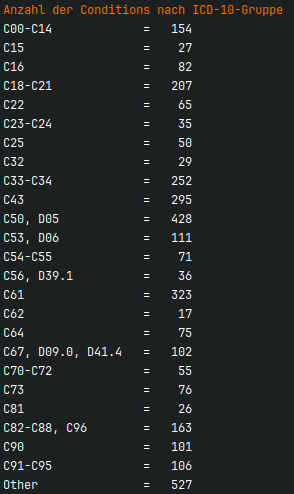
Die Anwendung gibt für die möglichen Quellen der Kennzahlen die Anzahl der Conditions, gruppiert nach ICD-10 Gruppen, aus.
Kennzahlen aus der CSV-Datei
Vor Veröffentlichung der Daten der CSV-Datei in Opal kann die Anzahl der Conditions, gruppiert nach ICD-10 Gruppen,
mit dem Befehl opal-file aus der CSV-Datei gewonnen werden.
bzkf-rwdp-check opal-file --file <Opal-CSV-Datei>.csv
Die Anwendung gibt nun eine Liste der ICD-10-Gruppen mit Anzahl der Conditions aus.
Kennzahlen aus der Onkostar-Datenbank
Die Anzahl der Conditions, gruppiert nach ICD-10-Gruppe, kann auch mit dem Befehl database aus der Onkostar-Datenbank
abgerufen werden.
bzkf-rwdp-check database --user me --year 2024
Die Anwendung gibt auch hier eine Liste der ICD-10-Gruppen mit Anzahl der Conditions aus.
Dieser Befehl hat noch weitere Parameter:
Options:
-D, --database <DATABASE> Datenbank-Name [default: onkostar]
-h, --host <HOST> Datenbank-Host [default: localhost]
-P, --port <PORT> Datenbank-Host [default: 3306]
-p, --password <PASSWORD> Passwort. Wenn nicht angegeben, wird danach gefragt
-u, --user <USER> Benutzername
-y, --year <YEAR> Jahr der Diagnose
Der zusätzliche Parameter --ignore-exports-since ist optional.
Wird er angegeben, werden keine Einträge mit Exportdatum ab diesem Datum verwendet.
Dies eignet sich um nachträglich Zahlen zu einem bestimmten Datum zu ermitteln.
Export aus der Onkostar-Datenbank
Die Anwendung ist in der Lage, mit dem Befehl export die Spalten
pat_id: Patienten-ID (optional über Parameter--pat-id)cond_id: Condition-IDconditiondate: Datum der Diagnosecondcodingcode: Der ICD-10-Code der Diagnose
in eine CSV-Datei zum Abgleich mit der OPAL-CSV-Datei zu exportieren.
Hierbei gelten die gleichen Datenbank-Parameter wie unter Kennzahlen aus der Onkostar-Datenbank, zusätzlich gibt es noch die folgenden Parameter:
Options:
--pat-id Export mit Klartext-Patienten-ID
-o, --output <OUTPUT> Ausgabedatei
Vergleich CSV-Datei für OPAL und Onkostar-Datenbank
Die Anwendung kann auch die Conditions in der CSV-Datei mit der Onkostar-Datenbank direkt vergleichen.
Hierzu kann der Befehl compare genutzt werden. Dieser verwendet alle Optionen für die Datenbank und die Option --file
für die CSV-Datei und gibt eine Übersicht auf der Konsole aus.